ゲノム解析とは?
ゲノム解析とは
ゲノム解析とは、生物のゲノムのもつ遺伝情報を総合的に解析することです。ゲノム解析は、ゲノムを構成するDNA分子の塩基配列(GATCのならび)を決めることから始まります。しかし、塩基配列データからだけでは、どこにどのような遺伝子があるのかは簡単にはわかりません。そこで、転写・翻訳によって作られるメッセンジャーRNAやタンパク質などの遺伝子産物の解析、生物種間で塩基配列がどれだけ似ているかなどの比較、さらに大腸菌や出芽酵母などの実験生物で解析された個々の遺伝子に関するデータなどを基に解析を進めます。
ゲノム解析では時に10億以上にもつながった塩基の配列をいろいろな観点から解析する必要がありますのでコンピュータの使用が不可欠です。コンピュータによってゲノムデータをはじめとする生物情報を解析する分野をバイオインフォマティクスと呼びます。
ゲノム塩基配列決定の流れ
NITEでは、生物の特徴を解析する上で必須となるゲノムの全塩基配列を解析し、得られた配列情報から遺伝子の同定と機能の推定を行って、その情報を一般に公開しています。塩基配列の決定は、大きく分けて(1)ゲノムDNAの切断と断片のクローン化、(2)各クローンの塩基配列(シーケンス)決定、(3)遺伝子領域と機能の推定、(4)論文発表とデータの公開、という手順を経て行われます。
(1)ゲノムDNAの切断と断片のクローン化
ゲノムDNAの抽出
図1-1
生物の細胞から糖、タンパク質、脂質等を除去し、ゲノムDNAを抽出します(図1-1)。
ショットガンクローンの作製
抽出したゲノムDNAは、そのまま塩基配列を解析するには大きすぎるため、まず以下の手順で適当な大きさに切断し、断片をランダムにクローン化します。
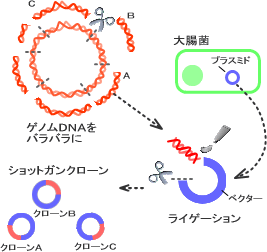
図1-2
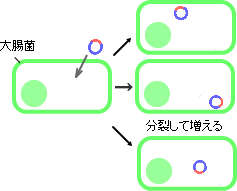
図1-3
それぞれのDNA断片を組み込んだプラスミドは大腸菌で増やすことができます(これを「クローン化する」と言います)。得られるクローンは、いろいろなゲノム断片をランダムに含んだものであることから「ショットガンクローン」と呼ばれます(図1-3)。
(2)各クローンの塩基配列(シーケンス)決定
シーケンス反応と塩基配列の読みとり
塩基配列の決定は、シーケンサと呼ばれる機械を用いて行います。シーケンサで塩基配列を読みとるために、解析対象のDNAに蛍光物質を付けます。ここでは、その操作をシーケンス反応と呼ぶことにします(図2-1)。
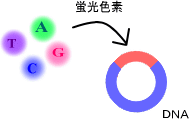
図2-1
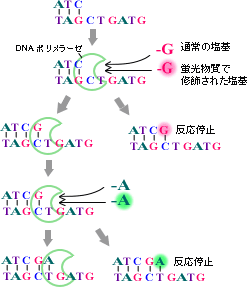
図2-2
まず、第一段階のシーケンス反応は、DNAポリメラーゼを使ってDNAを複製させることによって行います。DNAポリメラーゼは、1本鎖のDNAの「鋳型」のそれぞれのヌクレオチドに相補的なヌクレオチドを取り込んで新しいDNA鎖を作る酵素です。この時、通常のデオキシリボヌクレオチド(Deoxyribonucleic acid; dA、dG、dC、dT)に、それ以上DNA鎖が伸長できないジデオキシリボヌクレオチド(Dideoxyribonucleic acid; ddA、ddG、ddC、ddT)を加え、反応を行います。これらのジデオキシヌクレオチドには、異なる色を発光する蛍光物質が付いています(図2-2)。
このようにすると、鋳型のそれぞれのヌクレオチドに対応する位置に相補的なジデオキシヌクレオチドがランダムに取り込まれますので、全体としては1塩基ずつ長さの異なる合成鎖のセットができます(図2-3)。

図2-3 1塩基ずつの長さの異なる合成鎖
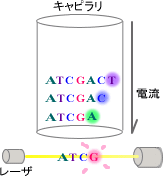
図2-4
このようにしてできた合成鎖のセットをDNAシーケンサのキャピラリ中で電気泳動(プラスとマイナスの電極の間でDNA分子を泳動させる)させます(図2-4)。DNAは、負に帯電しているので、合成鎖はすべて陽極へ泳動しますが、短いDNA鎖ほど早く、長いものほど時間がかかって遅く泳動します。そこで、キャピラリにレーザ光を当てて蛍光発色の時間(DNAの鎖の長さ)と蛍光の波長(塩基の種類)を検出するのです。これが塩基配列決定の概略です。

シーケンサ

キャピラリ
塩基配列データの処理
読みとった塩基配列データは数百から1,000塩基程度の長さの断片のデータです。上に述べたように、これらの配列データは、もともとゲノムDNAをランダムに切断して得られたショットガンクローンに由来するものですから、その多くは部分的に重複しています。そこでこれらのデータの重複をコンピュータで検出し、もとのゲノムDNAの塩基配列へと再構成していきます。この操作をアッセンブルと言います(図2-5)。
アッセンブルの過程では、ゲノムに散在するいろいろな種類の繰り返し配列の処理が問題になります。不確実な部分などがあった場合には、再度シーケンスを行うほか、アッセンブルの正しさを確認にするために、より大きな断片をもったクローンをシーケンスしてショットガンクローンのデータを位置づけたり、アッセンブルしたデータ間でPCRを行ったりして確認します。
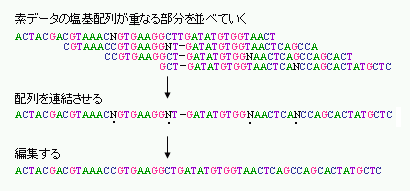
図2-5
塩基配列データの確認
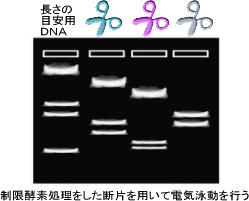
図2-6
再構成されたゲノムDNAの塩基配列の正確さを実験により確認する場合もあります。その場合には、塩基配列をデータを利用してゲノム上適当な間隔をおいてプライマーを作成し、ゲノムDNAをPCRによって増幅し、それを適当な制限酵素で切断(図2-6:ハサミの色は、制限酵素が異なることを示す)した後、電気泳動により切断パターンを確認します。

図2-7
これを塩基配列データから予想されるパターンと比較し、両者が一致すれば塩基配列データが正しく再構成されているとします(図2-7)。
(3)遺伝子領域と機能の推定
遺伝子領域の推定
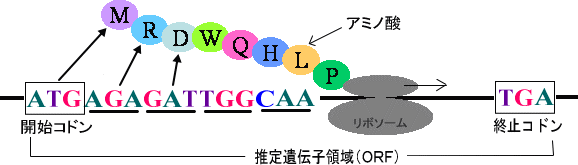
図3-1
塩基配列のうち、タンパク質として機能していると予想される領域をORF(Open Reading Frame:推定遺伝子領域)と呼びます。
ORFは、開始コドンと呼ばれる3塩基から始まり、終止コドンと呼ばれる3塩基で終わります。また、アミノ酸に対応するコドンも解読されており、ORFの推定はコンピュータを用いて行います。
推定遺伝子領域の機能推定
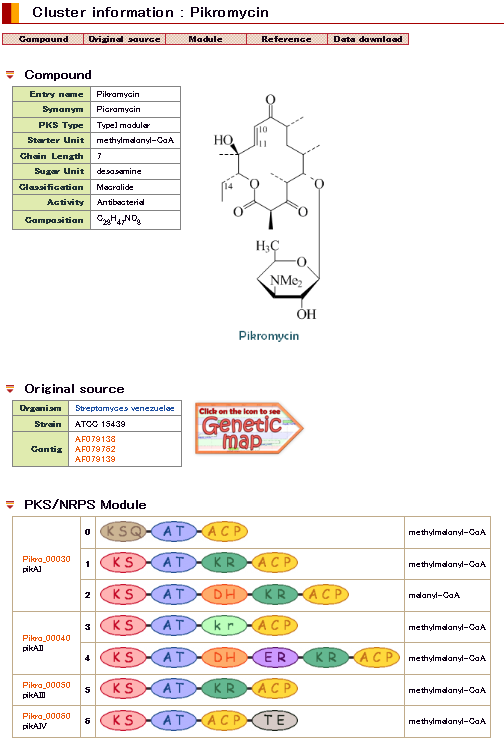
ORFをはじめとする塩基配列上の各種の単位の機能を推定し、意味づけすることを「アノテーション」と言います。NITEでは、主にホモロジー(相同性)検索(※3)やモチーフ検索(※4)等によってORFの機能を推定しています。この検索方法は、すでに研究された生物の遺伝子やタンパク質の機能に関するデータと、手持ちのデータを比較することにより、ORFの機能を推定する方法です。各ORFに対して「予測される機能」を示すことにより、データベースが研究者にとって使いやすいものとなります。
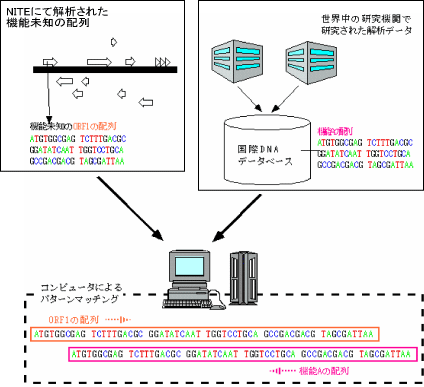
図3-2
(4)論文発表とデータ公開
塩基配列等の得られたデータを、国際的なデータベースである日本DNAデータバンク(DDBJ)に登録します。また、それらに考察を加え、論文を執筆し、学術誌へ投稿します。
NITEバイオテクノロジーセンター(NBRC)では、ホームページを通じてデータの公開を行っています。
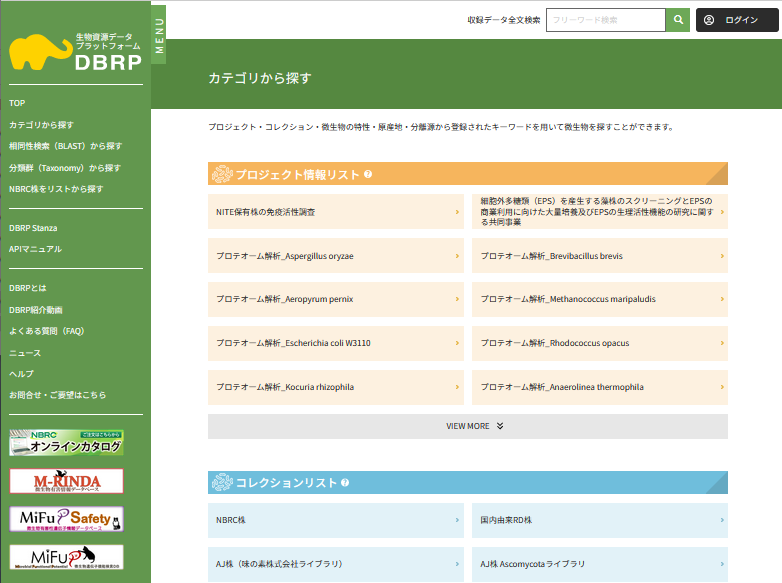
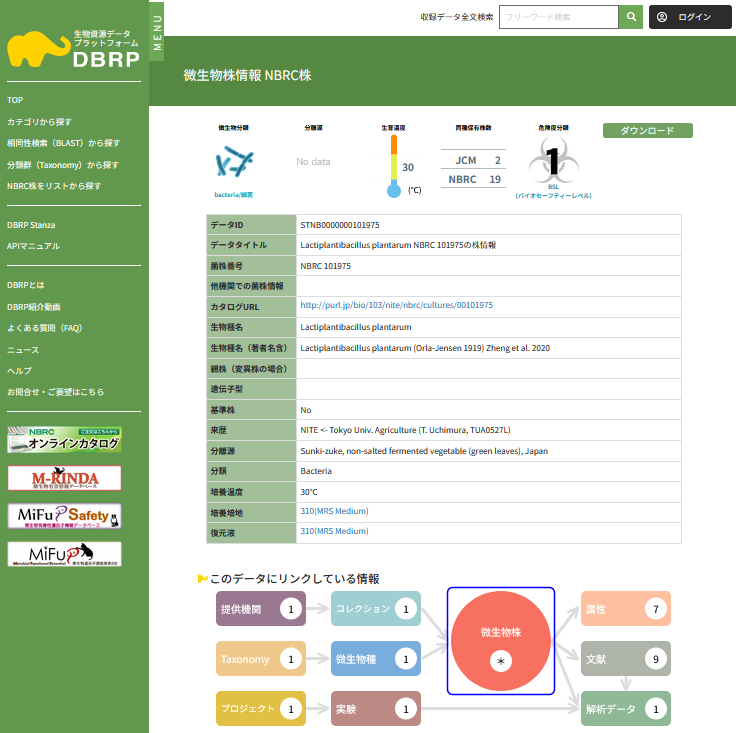
DBRPにおける生物資源情報の公開 (画像をクリックすれば拡大できます)
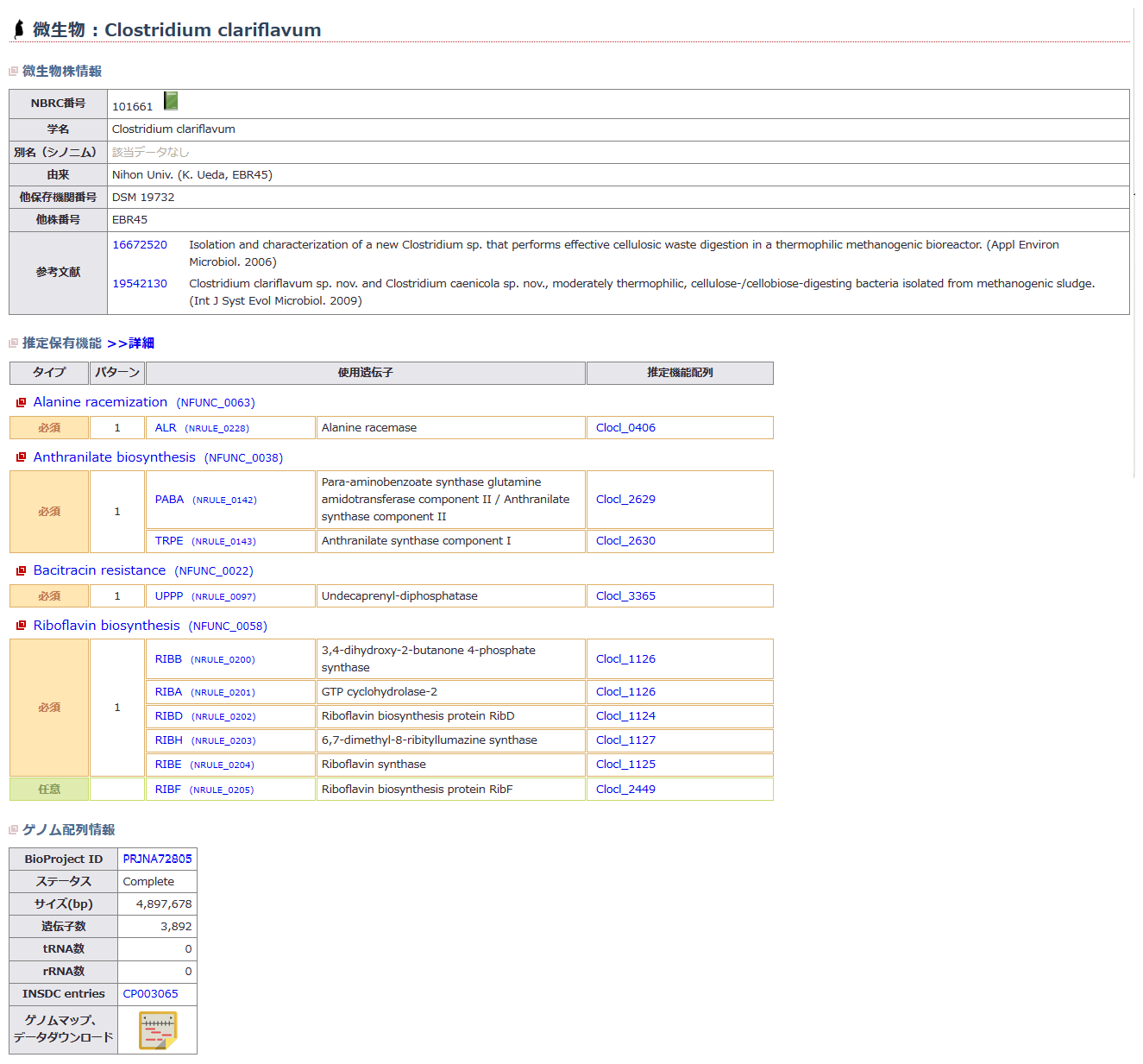
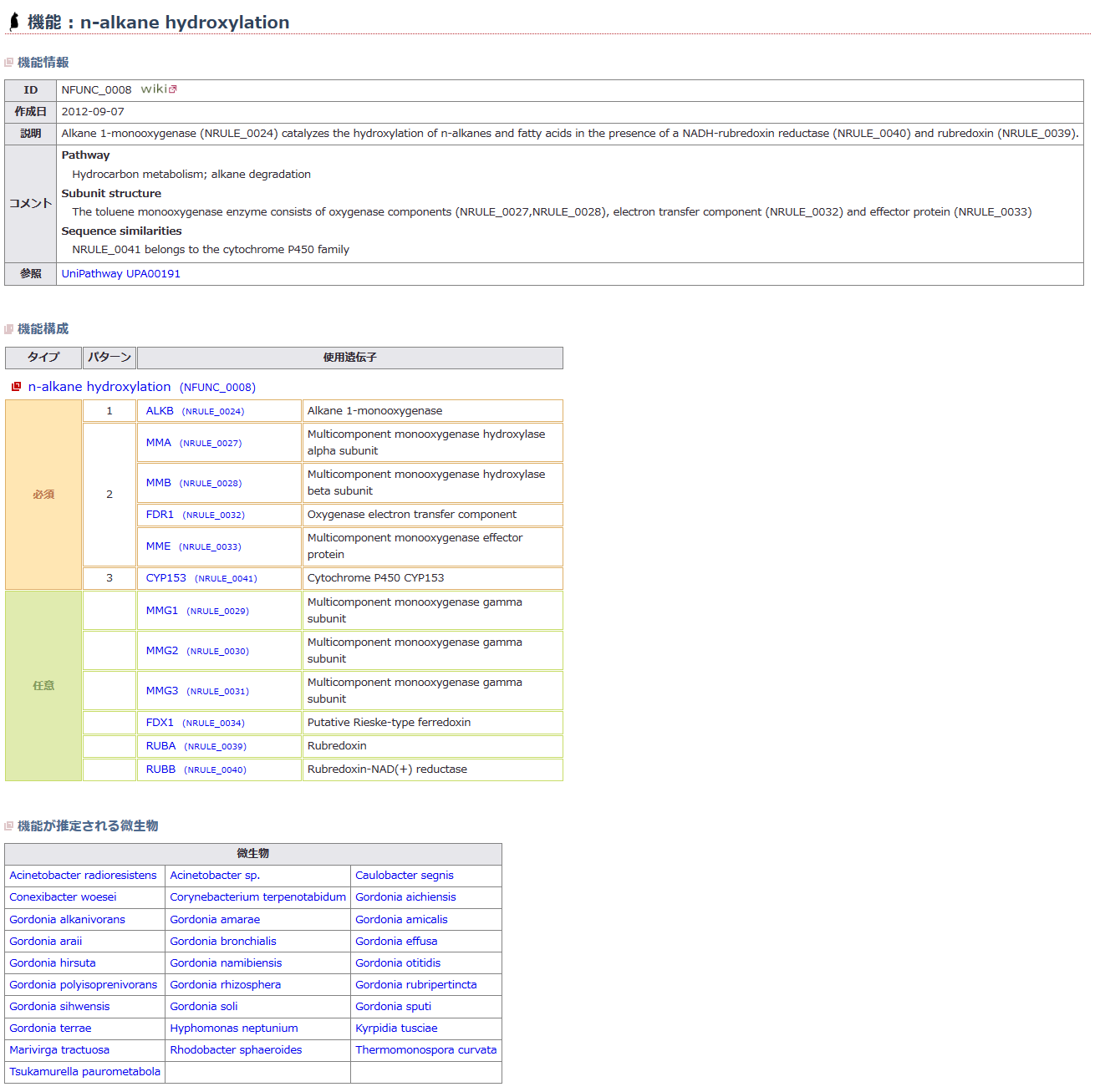
MiFuPにおける微生物遺伝子機能情報の公開 (画像をクリックすれば拡大できます)
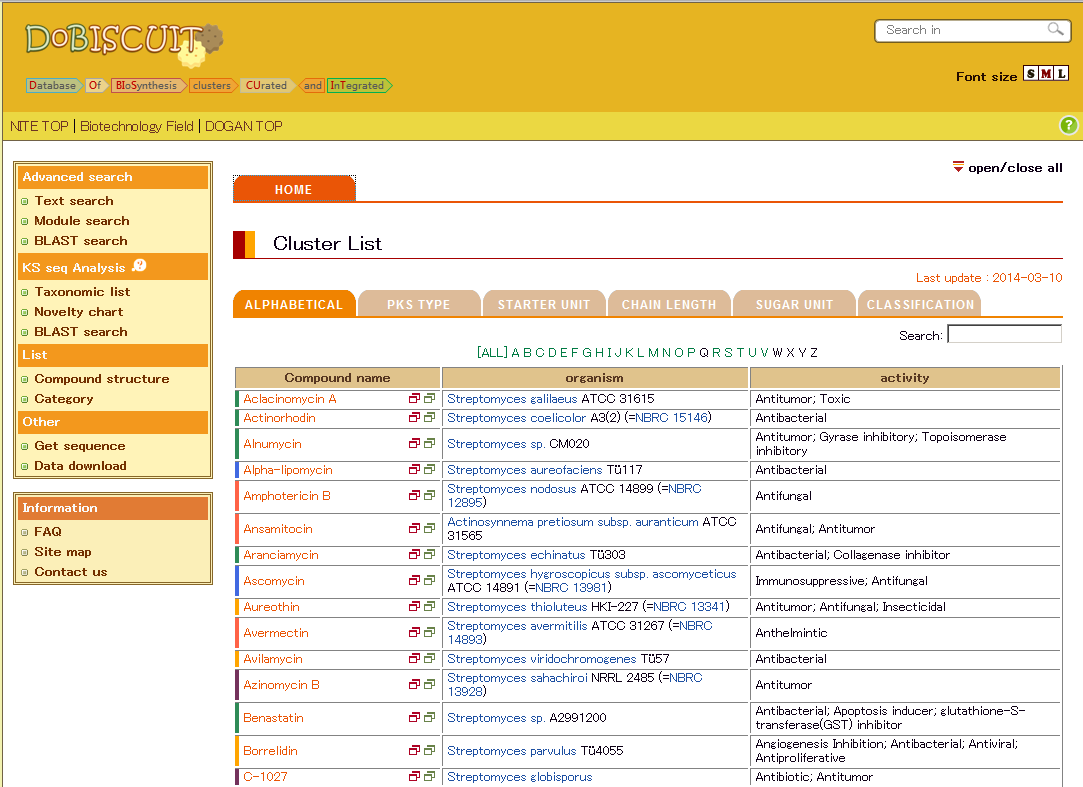
DoBISCUITにおける二次代謝産物合成遺伝子情報の公開 (画像をクリックすれば拡大できます)
お問い合わせ
- 独立行政法人製品評価技術基盤機構 バイオテクノロジーセンター 計画課
-
TEL:03-3481-1933
住所:〒151-0066 東京都渋谷区西原2-49-10 地図
お問い合わせフォームへ